2023.04.06
【プレスリリース】転写が盛んなゲノム領域も「塊」を作っていた!



概要
ヒトのゲノムDNAはクロマチンの形状によって、主に「ユークロマチン」「ヘテロクロマチン」の2つの領域に分類できるとされています。これまで長い間、頻繁に遺伝情報の読み出しが行われるユークロマチンのDNAはほどけている一方、遺伝情報の読み出しが抑えられているヘテロクロマチンのDNAは凝縮して塊を形成している、と考えられてきました。
このたび、情報・システム研究機構 国立遺伝学研究所 野崎慎 大学院生 (現 ハーバード大研究員)、井手聖助教、前島一博教授のグループ、東光一助教、黒川顕教授のグループは、理化学研究所 新海創也 上級研究員、大浪修一チームリーダーと共同で、生きた細胞内をナノメートル(1)レベルで可視化できる超解像蛍光顕微鏡(2)を用い、細胞の中でのDNAの動きを観察・解析し、ユークロマチンの挙動を詳細に調べました。
その結果、ユークロマチンにおいても、DNAが不規則に凝縮した「塊」を形成し、そのなかでDNAが揺らいでいることを発見しました。このことは、ユークロマチンにおいてはDNAがほどけている、という従来の定説を覆す発見で、不規則に凝縮した「塊」が、生きた細胞内におけるユークロマチンの基本構造であることがわかりました。また、ユークロマチンにおけるDNAの塊は、放射線などによるDNAの損傷への耐性にも貢献すると考えられます。
本研究の結果によって、生命の設計図であるDNAの遺伝情報がどのように維持され、どのように読み出されるのかについての理解が進むとともに、遺伝情報の保護、読み出し方の変化によって起きるさまざまな細胞の異常や関連疾患の理解につながることが期待されます。
成果掲載誌
本研究成果は、国際科学雑誌「Science Advances」に2023年4月6日(日本時間)に掲載されます。
論文タイトル: Condensed but liquid-like domain organization of active chromatin regions in living human cells
(ヒト生細胞のアクティブなクロマチン領域は凝集した液体様のドメインを形成する)
著者: Tadasu Nozaki*, Soya Shinkai*, Satoru Ide*, Koichi Higashi*, Sachiko Tamura, Masa A. Shimazoe, Masaki Nakagawa, Yutaka Suzuki, Yasushi Okada, Masaki Sasai, Shuichi Onami, Ken Kurokawa, Shiori Iida, Kazuhiro Maeshima
*equal contribution
(野崎慎*、新海創也*、井手聖*、東光一*、田村佐知子、島添將誠、中川正基、鈴木穣、岡田康志、笹井理生、大浪修一、黒川顕、飯田史織、前島一博)
*等しい貢献をした著者
DOI:10.1126/sciadv.adf1488
研究の詳細
研究の背景
私たちの体を構成する細胞一つ一つには、全長2メートルにおよぶゲノムDNAが収められています。DNAは直径2nmの細い糸状の物質で、「ヒストン」という樽状のタンパク質に巻き付くことで「ヌクレオソーム」という構造を作ります。ヌクレオソームにさまざまなタンパク質が結合することでDNAはさらに折りたたまれ、「クロマチン」と呼ばれる形を取り、細胞の中の「核」に収納されます(図1A)。
国立遺伝学研究所の前島教授らは、2008年よりヌクレオソームが不規則に、そしてダイナミックに細胞内に収められていることを提唱してきました(図1A中段)。
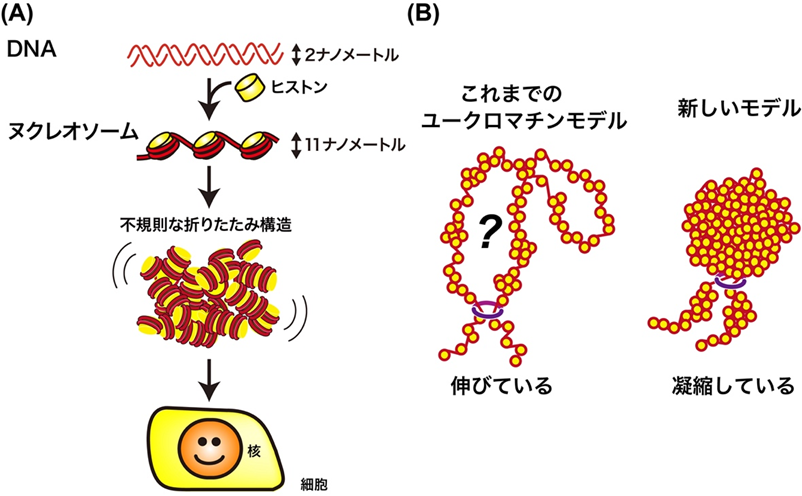
ヒトを含む真核生物のクロマチン(DNA)は、ユークロマチンもしくはヘテロクロマチンと呼ばれる2つの領域に分類されます(図1B)。ユークロマチンは、ヘテロクロマチンと比べてクロマチン(DNA)の密度が低い領域であり、遺伝情報を読み出す「転写」(3)が頻繁に行われています。これまで、ユークロマチンはヌクレオソーム同士がくっつかずに伸びたような(開いた)構造をとっていると考えられてきました。しかしながら、生きた細胞において、ユークロマチンをナノメートルのスケールで詳細に観察することは困難であったため、ユークロマチンが局所的にどんな構造をとっているのかは、明らかになっていませんでした。
本研究の成果
本研究では、まずヒトゲノム上のユークロマチンを特異的に蛍光標識する技術を開発し、標識の特異性を次世代DNAシークエンサーで確認しました。そしてヌクレオソーム1個1個を観察できる超解像蛍光顕微鏡を駆使し、蛍光標識したユークロマチンにおけるヌクレオソームの動きを生きた細胞において観察しました。さらに、2色の蛍光色素を用いてユークロマチン内の近接した2つのヌクレオソームの動きを同時に観察・比較したところ、150ナノメートル以内に近接した2つのヌクレオソームの動きが似ている(相関がある)ことがわかりました(動画)。このことから、ユークロマチンが、平均直径150ナノメートルほどの不規則に凝縮した「塊」(クロマチンドメイン)を形成していることが明らかとなりました(図2、吹き出し)。また、さらなる解析により、ユークロマチンにおいてヌクレオソームはクロマチンドメインの内部で液体のように動いていること、クロマチンドメインの数十倍程度のより大きなスケールでは、ユークロマチンは動かず、固体のように振る舞うこともわかりました(図2)。
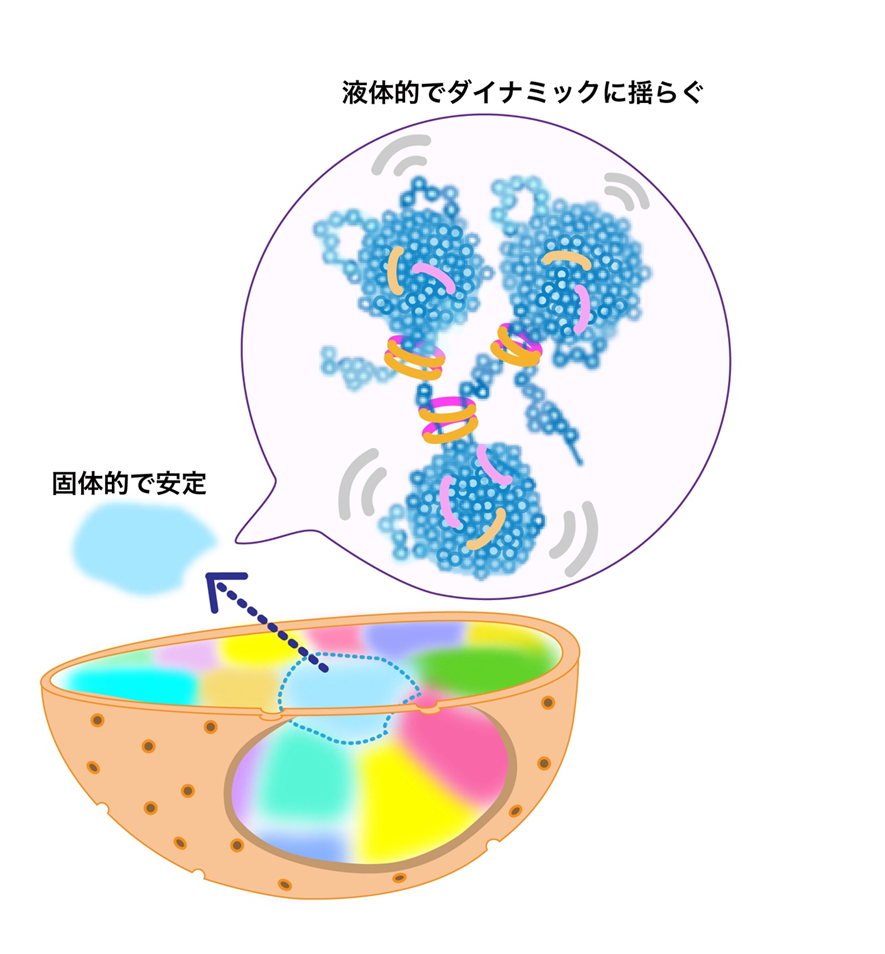
図2:ヌクレオソームはお互いにぺたぺたとくっついて、不規則に凝縮した「塊」を作っている(吹き出し中の3つの青い塊)。「塊」の中ではヌクレオソームはダイナミックに動き、液体のように振る舞うことによりDNA上で起こる反応を促進する。一方、より大きなスケールでは固体のように振る舞い、がんの原因になる長いクロマチンの絡まり・切断を防ぎ、遺伝情報の維持に貢献する。
これらの結果は生きた細胞を観察することができる顕微鏡により得られた新しいクロマチン像です。これまでの方法では細胞を固定(4)する必要があり、ヌクレオソームの動きや振る舞いの情報が失われてしまうという欠点がありました。今回明らかになった、小さいスケールでのユークロマチンの液体のような振る舞いは、タンパク質のドメイン内部への侵入やドメイン表面上のDNAの移動を促し、凝縮したクロマチンドメインにおける転写やDNA複製(5)、DNA修復(6)などの効率的な反応を可能にすると考えられます。また、ユークロマチンにおけるクロマチンの塊は、放射線などによるDNAの損傷への耐性にも貢献している可能性があります。さらに、より大きなスケールでのユークロマチンの固体のような振る舞いは、がんの原因となる長いクロマチンの絡まり・切断を防ぎ、遺伝情報の維持に貢献することが予想されます。
今後の期待
本研究では、転写が頻繁に行われている領域であるユークロマチンが、「塊」を作っていることを明らかにしました(図2)。この結果によって、細胞内で、転写に必要な因子がどのようにターゲット遺伝子を見つけるのか、どの遺伝子をいつ読み出すのか、といった転写の制御について、新しい描像が得られます。また、今回明らかになった小さいスケールでのクロマチンの液体のような振る舞いは、効率的なDNAの転写・複製・修復に直結し、より大きなスケールでの固体のような振る舞いは、遺伝情報の保持・維持につながると考えられます。このことから、本研究はこれらのクロマチンの振る舞いの変化が引き起こす異常や関連疾患の理解につながることが期待されます。
超解像蛍光顕微鏡により観察された生きた細胞の核内におけるヌクレオソームのゆらぎの動画。2色の蛍光色素を用いることで、近接したヌクレオソーム、あるいは離れたヌクレオソームを同時に観察できる。 上段のヌクレオソームの近接したペアは動きに相関が見られる。一方、下段の離れたヌクレオソームのペアは動きに相関が見られない。

用語解説
(1) ナノメートル
1メートルの10の9乗分の1(10-9)。
(2) 超解像蛍光顕微鏡
通常の光(可視光)を用いた顕微鏡で観察する場合は、200ナノメートル程度の大きさのモノをとらえるのが限界である(光の回折限界)。しかし、超解像蛍光顕微鏡はこの限界を超えて(超解像)、より小さな構造まで観察することができる。本研究では、クロマチンのヌクレオソームをまばらに蛍光標識することで超解像を達成する方法を用いた。
(3) 転写
DNAに書かれた、細胞の形態や機能を制御する分子の設計図、すなわち遺伝子を見つけ出し、その情報を読み出す反応のこと。読み出された情報をもとに、生命機能を司る特定の分子がつくられる。
(4) 固定
細胞の内部の構造を観察するために安定化(動かなく)すること。化学物質を用いる方法や、細胞を急速凍結する方法などがある。
(5) DNA複製
細胞が分裂する前に、遺伝情報を2つの娘細胞に引き継ぐために、細胞が持っている1セットのゲノムDNAをコピーして二倍に増やすこと。
(6) DNA修復
DNAが何らかの要因によって切断されたり、DNAの二本鎖がうまく噛み合わなかったりする際に、DNAにかかれた遺伝情報を元通りに直すこと。
研究体制と支援
本研究成果は、国立遺伝学研究所・ゲノムダイナミクス研究室の野崎慎 大学院生(遺伝研特別共同利用研究員、元学振特別研究員PD、現在 米ハーバード大学研究員)、井手聖 助教、田村佐知子 テクニカルスタッフ、島添將誠 総研大大学院生、飯田史織 総研大大学院生(元SOKENDAI特別研究員、現 学振特別研究員DC2)、前島一博 教授 、理化学研究所・生命機能科学研究センターの新海創也 上級研究員、大浪修一 チームリーダー、国立遺伝学研究所・ゲノム進化研究室の東光一 助教、黒川顕 教授、福岡工業大学の中川正基 准教授、東京大学の鈴木穣 教授、理化学研究所・生命機能科学研究センターの岡田康志 チームリーダー、京都大学の笹井理生 研究員との共同研究成果です。
本研究は、日本学術振興会(JSPS) 科研費(21H02453, 22H05606, 21H02535)、学術変革領域A「ゲノムモダリティ」(20H05936, 20H05550)、先進ゲノム支援(16H06279(PAGS), 22H04925(PAGS))、科学技術振興機構(JST) 戦略的創造研究推進事業(CREST) (JPMJCR15G2)、JST 次世代研究者挑戦的研究プログラム(JPMJSP2104)、上原記念生命科学財団の支援を受けました。
